cours 3 : langage R et RStudio
Mesures et tests d’association
Tableau de contingence et test du chi-deux
La commande table() permet de construire un tableau d’effectifs pour une variable qualitative, ou pour le croisement des modalités de deux variables qualitatives. Un tableau de contingence pour les variables Sexe et instruction peut être construit à l’aide de l’instruction suivante, en se rappelant que la première variable mentionnée dans la commande sera celle dont les modalités apparaîtront sous forme de lignes dans le tableau.
![]()
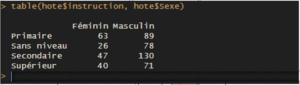
Si l’on stocke ce tableau de contingence dans une variable auxiliaire, tab, il est ensuite possible d’utiliser d’autres commandes pour travailler avec ce tableau, par exemple prop.table() discutée plus haut. Dans le cas d’un tableau à deux entrées, les fréquences relatives affichées sont calculées par rapport à l’effectif total.
Tableau de contingence et test du chi-deux
![]()
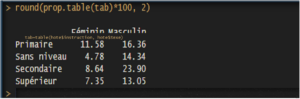
Il est possible de spécifier une option margin= pour indiquer à prop.table() comment calculer les fréquences relatives : avec margin = 1, R calcule les fréquences relatives conditionnellement aux totaux lignes (somme des effectifs par modalités de la première variable), tandis qu’avec margin = 2 les effectifs sont rapportés aux totaux colonnes.
Le test du chi-deux est disponible via la commande chisq.test(), et on lui fournit simplement un tableau de contingence. Il est également possible d’indiquer séparément les deux variables, par exemple : chisq.test(hote$instruction, hote$sexe).
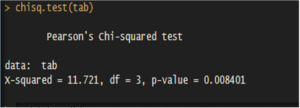
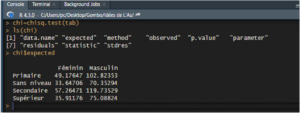
Si l’on stocke le résultat du test dans une variable auxiliaire, il est possible d’obtenir des informations supplémentaires comme, par exemple, les effectifs théoriques (effectifs attendus sous l’hypothèse d’indépendance entre les deux variables).
On procédera de la même manière que lorsque l’on souhaite accéder à une variable dans un data frame, c’est-à-dire en indiquant le nom de la variable suivi du symbole $ et du nom de l’objet d’intérêt, dans ce cas expected. Pour voir la liste des information supplémentaires qu’il est possible d’obtenir, il suffit d’appliquer la commande ls() à la variable auxiliaire qui contient les résultats du test.
La commande fisher.test() permet de faire le test de Fisher en suivant la même procédure.
Corrélation linéaire
Pour calculer le coefficient de corrélation linéaire de Pearson, on utilisera la commande cor(). Si les variables présentent des valeurs manquantes, il faudra explicitement demander à R d’éffectuer le calcul sur l’ensemble des paires de valeurs complètes à l’aide de l’option use = “pairwise“. Voici un exemple d’utilisation avec deux variables de type numérique.
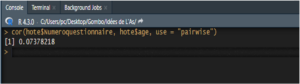
Le coefficient de corrélation de rangs de Spearman est quant à lui obtenu en ajoutant l’option method = “spearman’’ à la commande précédente.
La commande cor.test() renvoit en plus du test de nullité du coefficient de corrélation un intervalle de confiance (par défaut à 95 %) et la valeur du coefficient de corrélation.
Analyse de variance à un facteur
Si on s’intéresse à l’âge des hôtes dans les différentes régions. Pour calculer l’âge moyen dans chaque groupe, on peut utiliser aggregate() avec la commande mean() en troisième argument. De même, il serait possible de vérifier la valeur des variances, ou n’importe quelle autre quantité, en adaptant la commande à appliquer à la variable âge selon les différentes régions.
![]()
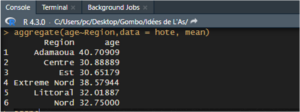
Analyse de variance à un facteur
Pour avoir une meilleure appréciation, une représentation graphique tel un diagramme de type boîte à moustaches permettra de visualiser la distribution des données dans chaque groupe. La commande boxplot() repose sur l’usage de la même formule que aggregate() permettant de décrire la relation entre les deux variables.
![]()
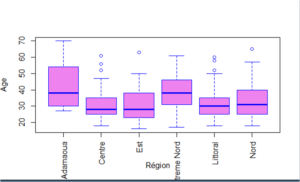
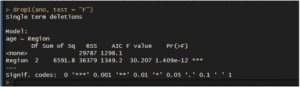
Pour réaliser le modèle d’analyse de variance (ANOVA) à un facteur, on utilisera directement la commande lm(). Cette fonction permet de décrire les variations de la variable réponse (ic, âge) en fonction de la variable explicative, de type facteur (ici, la région). Par ailleurs, lm() dispose d’une option subset=, ce qui permet de choisir les sous populations (ici, les régions) dans lesquelles on souhaite faire l’analyse de la variance.
![]()
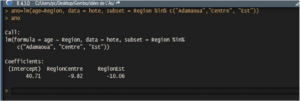
R retourne par défaut, un résultat incluant uniquement les coéfficients de régression calculés selon la méthode de codage par contrastes de traitement pour les modalités de la variable qualitative : chaque coéfficient représente la déviation moyenne par rapport à la catégorie de référence, dont la moyenne est représentée par le terme d’ordonnée à l’origine (“Intercept”), qui est toujours sous R le premier niveau du facteur (ici, “Adamaoua”). Pour obtenir le tableau d’ANOVA, on utilisera drop1() en précisant le type de test à réaliser, test = ‘’F’’ signifiant un test de Fisher-Snedecor. On peut aussi utiliser la commander anova().
Le langage R Markdown
R Markdown est un outil permettant de produire des rapports d’analyse détaillés et commentés, plutôt que de simples scripts R incluant quelques commentaires. Il offre donc une syntaxe simplifiée pour mettre en forme des documents contenant à la fois du texte, des instructions R et le résultat fourni par R lors de l’évaluation de ces instructions. La nouvelle implémentation R Markdown v2 est disponible depuis 2014 dans le logiciel Rstudio. Initialement, la possibilité de transformer du texte contenant des instructions R en document HTML ou PDF reposait sur les packages knitr et markdown, mais à présent il n’existe plus qu’un seul package qui se charge de gérer la conversion des documents R Markdown (extension. Rmdou .rmd) en documents PDF, HTML ou DOCX.
Markdown est un langage permettant de baliser du texte simple à l’aide de symboles prédéfinis, un peu à l’image des balises HTML, afin de produire une sortie enrichie avec des titres, des paragraphes, etc.
Il s’agit donc principalement de rédiger un document de type texte, dans un éditeur approprié (éviter MS Word), et d’insérer des symboles simples afin de signaler les mots à mettre en surbrillance ou la délimitation des titres de section, par exemple.
Pour la génération de rapports automatiques, les packages rmarkdown, markdown et knitr
doivent être installés.
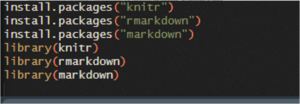
Mais Sur Rstudio, un seul Package suffit désormais pour prendre en charge la génération des rapports automatiques avec R markdown
install.packages(“pak”)
pak::pak(‘rstudio/rmarkdown’)
Ouvrez un nouveau fichier R markdown et enregistrez le
File –> New File –> Rmarkdown
Laissez toutes les options par défaut (document et html), et remplissez le champs “Title”, ce titre correspondra au titre du document, il peut être long.
Dans votre fichier Markdown, les titres de sections sont préfixés d’un ou plusieurs #, selon le niveau de profondeur du titre. Par exemple, pour un titre de niveau 1, on écrira # Titre de niveau 1, alors que pour un titre de niveau 2 on écrira ## Titre de niveau 2 (à ne pas confondre avec le symbole de commentaire des scripts R). Au niveau des éléments de texte, la mise en gras s’effectue en encadrant le texte par ** et la mise en italique par *. Pour utiliser une police à espacement fixe (monospace), on encadrera le texte avec des deux quotes simples inversées (‘).
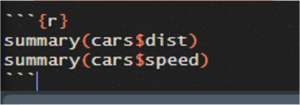
Les instructions R sont encadrées par “‘{r} et “‘ (appelé “code chunk”), comme ci-dessous :
Par ailleurs, le Menu « Help » de Rstudio propose un apperçu des différentes synthaxes de Rmarkdown.
La génération du document final se fait en cliquant sur le bouton Knit HTML(ou Knit PDF) selon l’option choisie). Dans le cas du format PDF, il est nécessaire d’avoir un système LaTeX (Tel MikTex) installé sur le système.
Références
Christophe Lalanne & Bruno Falissard, Introduction au langage R et à RStudio
Vincent Goulet, Introduction à la programmation en R, École d’actuariat, Université Laval
Grandud, C. (2013). Reproducible Research with R and RStudio.
Chapman & Hall/CRC 2. Xie, Y. (2013). Dynamic documents with R and knitr.

